IGV (Itegrative Genomics Viewer)是一款功能强大的综合性基因组学可视化工具,能够将基因组的变异情况进行可视化,因此广泛应用于基因组学的研究中。IGV有着良好的图形界面,在window系统上操作十分便利。然而,当我们需要批量处理生信数据时,Linux系统是一个更好的选择。不幸的是,IGV在Linux上的使用并不友好。本文记录了在Linux上使用IGV的具体方法。
环境配置
安装 IGV
不同版本的IGV依赖于不同版本的Java,因此我们需要先确定要安装的IGV版本,再安装对应版本的Java。
我们可以从IGV的Download页面下载IGV安装包。由于我们是在Linux中运行IGV,所以可以选择下载 Command line IGV and igvtools for all platforms (Separate Java 17 required) 到自己在服务器上的工作目录。解压安装包后直接运行安装包内的igv.sh文件即可运行IGV。
# 下载IGV安装包
wget https://data.broadinstitute.org/igv/projects/downloads/2.18/IGV_2.18.0.zip
# 解压IGV安装包
unzip IGV_2.18.0.zip
# 运行IGV(需要正确安装了指定版本的Java后才能正常运行)
cd IGV_2.18.0
igv.sh安装依赖
IGV的运行依赖于指定版本的Java。另外,使用IGV生成基因组可视化图的过程中还需要调用图形界面(X Window服务器)。在Linux中可以使用Xvbf (X Virtual Frame Buffer) 虚拟一个X Window服务器供IGV使用。最后,由于IGV直出的图还是不太符合我的需求,因此我还安装了imagemagick,用来裁剪图片。
创建 Singularity 容器
由于IGV指定了Java版本,不利于我们在Linux中进行版本管理。因此本文中使用了Singualrity创建了一个独立的Linux容器供IGV使用。这样做的另一个好处就是可以很方便的实现系统迁移。只要把这个Linux容器复制到另一个安装了Singularity的Linux系统中,就可以直接在新的Linux系统中运行IGV。
Singularity是劳伦斯伯克利国家实验室专门为大规模、跨节点HPC和DL工作负载而开发的容器化技术,具备轻量级、快速部署、方便迁移等优势,且支持从Docker镜像格式转换为Singularity镜像格式。
用不用Singularity都可以,本文使用只是方便我管理运行环境而已。Singularity的安装可以参考《Singularity 安装与使用教程:高性能计算环境容器化指南》
1. 下载Singularity基础容器
首先需要一个Linux基础容器 (base images)。可以去这个页面下载官方的Linux基础容器,也可以用下方的命令直接拉取一个合适的基础容器:
# 从Singularity拉取一个基础Ubuntu容器
[root@comput1 ~/example]$ singularity pull library://library/default/ubuntu
INFO: Downloading library image
28.44 MiB / 28.44 MiB [================================] 100.00% 2.66 MiB/s 10s
WARNING: unable to verify container: ubuntu_latest.sif
WARNING: Skipping container verification
[root@comput1 ~/example]$ ls
ubuntu_latest.sif2. 创建Singularity沙盒
Singularity拉取下来的容器是一个只读格式的sif文件。为了在里面安装软件,需要将其转换成一个沙盒类型的文件夹(IGV_env):
# 将sif文件转换为沙盒
[root@comput1 ~/example]$ singularity build --sandbox IGV_env/ ubuntu_latest.sif
INFO: Starting build...
INFO: Creating sandbox directory...
INFO: Build complete: IGV_env/
[root@comput1 ~/example]$ ls
IGV_env ubuntu_latest.sif3. 配置沙盒内环境
现在我们可以进去沙盒,并在其内部安装我们需要的软件:
# 以可写方式进入沙盒
[root@comput1 ~/example]$ singularity shell --writable IGV_env/
Singularity>
# 更新apt
Singularity> apt-get update
# 安装 Java 17(本次使用的 IGV 所指定的 Java 版本)
Singularity> apt install -y openjdk-17-jdk
# 安装 Xvfb
Singularity> apt-get install -y xvfb
# 安装 imagemagick
Singularity> apt-get install -y imagemagick
# 退出沙盒
Singularity> exit4. 打包沙盒为新的只读sif文件
配置好内部环境的沙盒可以重新打包为只读形式的sif文件,更加安全且方便移植:
# 将sif文件转换为沙盒
[root@comput1 ~/example]$ singularity build IGV_env.sif IGV_env/
INFO: Starting build...
INFO: Creating SIF file...
INFO: Build complete: IGV_env.sif
[root@comput1 ~/example]$ ls
IGV_env IGV_env.sif ubuntu_latest.sif如果你选择按照上述方式成功配置了一个Singularity容器,那你现在就可以跳转到生成基因组可视化图部分。如果你在Singularity内安装软件时出现问题,可以参考下面几个小章节的内容。如果你不打算使用Singularity,那么你就需要参考下面几个小章节的内容,在你的Linux系统上安装好IGV需要的软件。
安装 Java
1. 使用包管理器安装Java
IGV的运行依赖于指定版本的Java,我们需要根据自己的IGV版本安装对应版本的Java。我们可以使用以下命令在Linux上安装Java:
apt-get update
# 查看apt提供了的Java版本
apt search openjdk | grep -E 'openjdk-.*-jdk/'
# 本文中安装的是Java17
apt install -y openjdk-17-jdk
# 查看Java版本
java --version2. 手动安装Java
# 下载Java安装包
wget https://download.oracle.com/java/17/latest/jdk-17_linux-x64_bin.tar.gz
# 新建一个安装路径
mkdir /etc/java-17-openjdk/
# 将Java安装包解压到安装路径
tar -xvf jdk-17_linux-x64_bin.tar.gz -C /etc/java-17-openjdk/
# 将Java添加进环境变量(临时)
export JAVA_HOME=/etc/java-17-openjdk/jdk-17.0.12/
export PATH=$PATH:$JAVA_HOME/bin安装 Xvbf
使用IGV生成基因组可视化图的时候需要调用图形界面,但是Linux默认是不提供图形界面的,因此我们使用IGV生成可视化图的时候会报错。遇到这种情况时,我们一般不会特地去安装一个图形界面,而是选择使用Xvfb来创建一个虚拟图形界面供IGV使用。
Xvfb(X Virtual Frame Buffer)是基于X Window的虚拟显示服务器,可以在没有物理显卡图形界面和人机交互界面的情况下,运行X Window相关应用程序。它模拟了一个完整的X Window系统,包括显示和输入设备,但一切都是在内存中进行处理,没有显示器和键盘。这使得测试和调试GUI应用程序变得更加容易和自动化。
我们可以使用以下命令安装Xvfb:
# 安装Xvfb
apt-get install -y Xvfb
# 运行Xvfb (Ctrl+C关闭服务)
Xvfb实际应用时,我们可以使用Xvfb提供的一个shell脚本调用Xvfb。xvfb-run是Xvfb提供的一个Shell脚本,它可以自动启动和停止 Xvfb ,并将 DISPLAY 环境变量设为正确的值,使用它可以简化启动过程的步骤。使用时只需在需要启动的命令前加上 xvfb-run:
# 使用xvfb-run启动需要调用图形界面的软件
xvfb-run -a --server-args="-screen 0 1280x800x24 -nolisten tcp" igv.sh --batch Batch_scripts_example.txt安装 Imagemagick
可能是我还没完全摸透,或者IGV本身存在一些bug,总之IGV出的可视化图并没有完全满足我的需求。因此本文中还安装了Imagemagick,用于对IGV绘制的可视化图进行一定程度的裁剪。
Imagemagick是一款强大的图像处理软件,它可以用于创建、编辑、合并和转换图像。它支持超过200种图像格式,并且提供了丰富的功能,包括图像缩放、旋转、裁剪、加水印、添加特效等。ImageMagick还支持批量处理图像,可以通过命令行或者编程接口进行调用。它是一款开源软件,可以在多个操作系统上运行,包括Windows、Linux、Mac等。
使用以下命令安装Imagemagick:
# 安装 imagemagick
apt-get install -y imagemagick
# 裁剪图片 (-crop后的参数为:图片宽度x图片高度+起始位点横坐标+起始位点纵坐标。其中图片左上角的横纵坐标为(0,0))
convert example.raw.png -crop 968x485+167+0 example.png切换 apt 源
由于国内的网络原因,使用apt安装上述软件有可能会失败。这个时候可以考虑使用国内镜像替换掉apt的源:
这个方法不适用于Java,国内的镜像提供的Java版本太少了,可以试试手动安装Java。不过另外两个软件安装失败时可以尝试这种方法。
# 备份源文件
cp /etc/apt/sources.list /etc/apt/sources.list.bak
# 切换源,下面三个源任选一个执行
## 中科大源
echo '
# 默认注释了源码仓库,如有需要可自行取消注释
deb https://mirrors.ustc.edu.cn/ubuntu/ focal main restricted universe multiverse
# deb-src https://mirrors.ustc.edu.cn/ubuntu/ focal main restricted universe multiverse
deb https://mirrors.ustc.edu.cn/ubuntu/ focal-security main restricted universe multiverse
# deb-src https://mirrors.ustc.edu.cn/ubuntu/ focal-security main restricted universe multiverse
deb https://mirrors.ustc.edu.cn/ubuntu/ focal-updates main restricted universe multiverse
# deb-src https://mirrors.ustc.edu.cn/ubuntu/ focal-updates main restricted universe multiverse
deb https://mirrors.ustc.edu.cn/ubuntu/ focal-backports main restricted universe multiverse
# deb-src https://mirrors.ustc.edu.cn/ubuntu/ focal-backports main restricted universe multiverse
# 预发布软件源,不建议启用
# deb https://mirrors.ustc.edu.cn/ubuntu/ focal-proposed main restricted universe multiverse
# deb-src https://mirrors.ustc.edu.cn/ubuntu/ focal-proposed main restricted universe multivers
' > /etc/apt/sources.list
## 更新源
apt-get update
## 阿里源
echo '
deb http://mirrors.aliyun.com/ubuntu/ xenial main
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial main
deb http://mirrors.aliyun.com/ubuntu/ xenial-updates main
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial-updates main
deb http://mirrors.aliyun.com/ubuntu/ xenial universe
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial universe
deb http://mirrors.aliyun.com/ubuntu/ xenial-updates universe
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial-updates universe
deb http://mirrors.aliyun.com/ubuntu/ xenial-security main
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial-security main
deb http://mirrors.aliyun.com/ubuntu/ xenial-security universe
# deb-src http://mirrors.aliyun.com/ubuntu/ xenial-security universe
' > /etc/apt/sources.list
## 更新源
apt-get update
## 清华源
echo '
# 默认注释了源码镜像以提高 apt update 速度,如有需要可自行取消注释
deb https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial main restricted universe multiverse
# deb-src https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial main restricted universe multiverse
deb https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-updates main restricted universe multiverse
# deb-src https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-updates main restricted universe multiverse
deb https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-backports main restricted universe multiverse
# deb-src https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-backports main restricted universe multiverse
deb https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-security main restricted universe multiverse
# deb-src https://mirrors.tuna.tsinghua.edu.cn/ubuntu/ xenial-security main restricted universe multiverse
' > /etc/apt/sources.list
## 更新源
apt-get update切换源之后再尝试使用apt安装需要的软件。
生成基因组可视化图
在Linux上使用IGV生成基因组可视化图的过程实际上是利用了IGV提供的 run batch script 功能。如果你在Windows系统上使用过IGV,就可以看出来 Batch Script 其实模拟了用户在图形界面中手动生成基因组可视化图的过程。用户的每一次操作都被简化成了一个命令语句。这一系列命令语句合并在一起形成了一个 Batch Script 文件,指导了IGV要如何生成基因组可视化图。
制作 Batch Script 文件
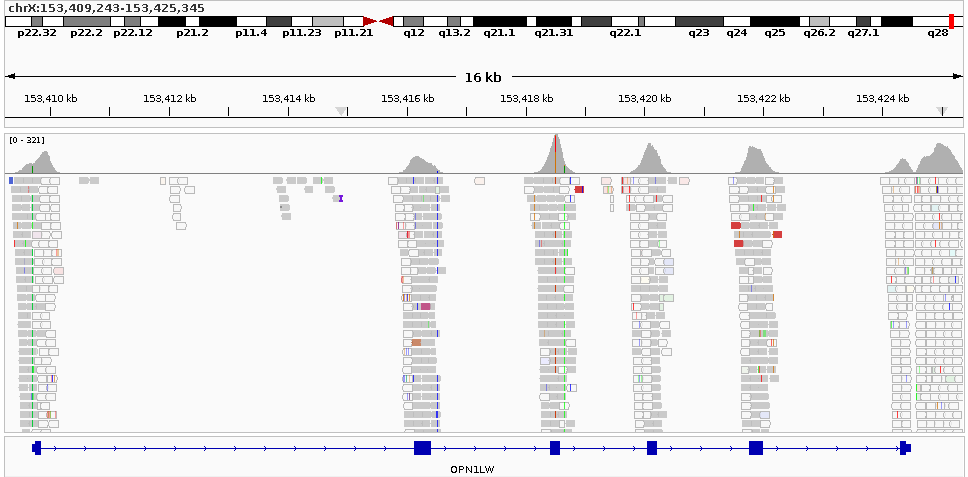
下面的 Batch Script 文件内容可以用于生成OPN1LW区域的基因组可视化图:
genome hg19
load ./testSample.deduped.bam name=testSample
goto chrX:153,409,243-153,425,345
sort position
collapse
maxPanelHeight 300
preference NAME_PANEL_WIDTH 0
snapshotDirectory ./mySnapshotDirectory
snapshot testSample.OPN1LW.raw.png
exit这里,IGV会自动下载并加载hg19的参考基因组信息,但是大多数情况下将参考基因组信息存储在本地服务器上是一个更稳定的选择。我们可以将IGV自动下载的参考基因组文件保存在本地服务器上,然后通过一个 .genome 文件指导IGV如何在本地服务器上加载参考基因组文件。
IGV自动下载hg19的参考基因组文件时,会给出一个
.json文件的超链接。可以将这个文件用wget下载下来,里面有hg19所需要的参考基因组文件的超链接。可以使用这些超链接把基因组文件下载到本地。
目前我还没有找到在linux上制作 .genome 文件的方法,因此这一步是在Windows下完成的。首先安装Windows版的IGV,在IGV的Download页面下载并安装 IGV for Windows (Java included) 即可。然后把本地服务器上下载的参考基因组信息传到Windows上,其中基因组序列文件是必要的,其他为可选,如CytoBand和alias文件。接下来打开IGV,在菜单栏中依次点击 Genomes -> Create .genome file… 。填写好基因组信息和文件路径后点击OK(Gene空着即可,把CytoBand和alias填上就行,当然,不填也无所谓,只有序列文件是必选项)。最后选择 .genome 文件的保存位置即可。这里要注意一下,最好把 .genome 文件和参考基因组文件全都保到同一路径下,这样 .genome 文件就会直接记录参考基因组文件的不含路径信息的文件名,否则,会记录进去一部分路径信息,不利于我们将 .genome 文件上传到Linux中使用。通过如上方式制作的 .genome 文件会在 .genome 文件所在的目录下寻找基因组信息文件,因此把 .genome 文件上传到Linux中存放基因组信息文件的路径下后,IGV就可以通过 .genome 文件索引到对应的基因组信息文件。
为了加载本地的 .genome 文件,我们需要修改一下 Batch Script 文件。值得注意的是,我们需要单独加载一下 ncbiRefSeq.txt.gz 文件,这个是基因组可视化图最下方的基因信息。因为我没有搞明白生成 .genome 文件时需要的 Gene 文件是何种格式,所以最后以这种方式手动添加了基因文件:
genome ./genome/hg19/hg19.genome
load ./genome/hg19/ncbiRefSeq.txt.gz name=Gene
load ./testSample.deduped.bam name=testSample
goto chrX:153,409,243-153,425,345
sort position
collapse
maxPanelHeight 300
preference NAME_PANEL_WIDTH 0
snapshotDirectory ./mySnapshotDirectory
snapshot testSample.OPN1LW.raw.png
exit运行 IGV
可以制作一个shell文件(run.sh),便于我们使用前面制作的Singularity容器运行IGV:
# 如果使用了手动方式安装的Java,则取消掉下面两行的注释,将Java添加到临时环境变量中
# export JAVA_HOME=/etc/java-17-openjdk/jdk-17.0.12/
# export PATH=$PATH:$JAVA_HOME/bin
# 使用Xvfb运行IGV生成IGV可视化图。IGV会根据batch文件中指定的命令生成基因组可视化图
xvfb-run -a --server-args="-screen 0 2560x720x24 -nolisten tcp" /root/shenh/singularity/IGV/IGV_2.18.0/igv.sh --batch ./Batch_script.txt
# 裁剪IGV生成的基因组可视化图
convert ./mySnapshotDirectory/testSample.OPN1LW.raw.png -crop 968x485+167+0 ./mySnapshotDirectory/testSample.OPN1LW.png实际上 Batch Script 文件中的 preference NAME_PANEL_WIDTH 0 命令就应该把可视化图最左侧的 Name Panel 栏删除了的,可是不知道为什么没有生效。最后不得不使用 Imagemagick 裁剪掉左侧区域。
运行以下命令生成基因组可视化图:
# 对于使用了Singularity的用户,命令如下
singularity exec IGV_env.sif sh run.sh
# 对于未使用Singularity的用户,命令如下
sh run.sh未解决的问题
1. 当我把制作好的IGV_env.sif文件移植到生产端的服务器上运行时,过程中总会产生如下错误:
(process:45461): dconf-CRITICAL **: 11:49:23.924: unable to create directory '/run/user/1019/dconf': Read-only file system. dconf will not work properly.目前来看,这个错误不影响出结果。尝试定位原因发现生产端服务器的Xvfb服务默认就已经被root用户开启了,而测试用的本地服务器只有在测试IGV是才会打开Xvfb。不知道是不是这个原因导致的上述错误。
# 在生产端服务器尝试运行Xvfb,发现已经被管理员运行了
Singularity> Xvfb
(EE)
Fatal server error:
(EE) Server is already active for display 0
If this server is no longer running, remove /tmp/.X0-lock
and start again.
(EE)
Singularity> ls -l /tmp/.X0-lock
-r--r--r--. 1 root root 11 May 27 19:18 /tmp/.X0-lock
发表回复