对bed文件的集合操作是生信分析中的重要组成部分,不过由于集合操作的效果往往五花八门,所有在这里做个最基本的记录。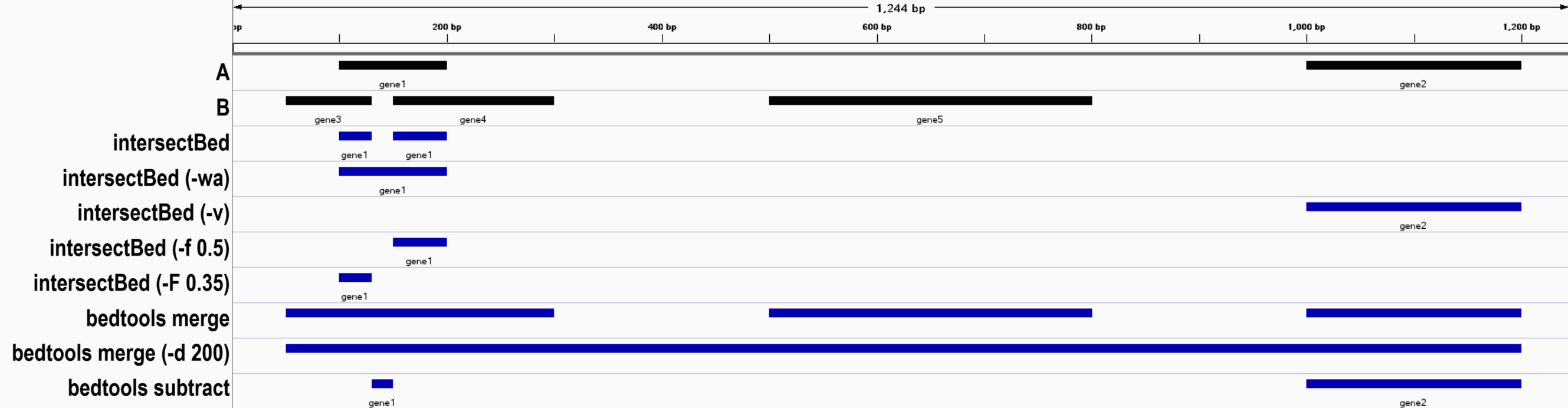
本文的测试环境为 bedtools(v2.31.1),并使用进程替换进行测试,这样就不用创建测试文件了,格式如下:
intersectBed -a <(echo -e "chr1\t100\t200") -b <(echo -e "chr1\t150\t300")
取交集(intersectBed 等价于 bedtools intersect)
默认操作,输出 A 中与 B 重叠的区间。
1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5")
# 结果如下
chr1 100 130 gene1
chr1 150 200 gene1保留 A 的完整原始行(即使部分重叠)
参数:-wa (write original A entry)1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -wa
# 结果如下
chr1 100 200 gene1
chr1 100 200 gene1输出 A 中与 B 重叠的区间,并追加的 B 的完整重叠行
参数:-wb1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -wb
# 结果如下
chr1 100 130 gene1 chr1 50 130 gene3
chr1 150 200 gene1 chr1 150 300 gene4保留 A 的完整原始行以及 B 中完整重叠行
参数:-wa -wb1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -wa -wb
# 结果如下
chr1 100 200 gene1 chr1 50 130 gene3
chr1 100 200 gene1 chr1 150 300 gene4保留 A 的完整原始行以及 B 中完整重叠行,并输出重叠部分长度
参数:-wo (write overlap)1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -wo
# 结果如下
chr1 100 200 gene1 chr1 50 130 gene3 30
chr1 100 200 gene1 chr1 150 300 gene4 50保留 A 的所有完整原始行以及 B 中完整重叠行,对于在 B 中不存在重叠行的 A,使用“-1”或“.”填充,在最后输出重叠部分长度
参数:-wao (Write All and Overlap)1
2
3
4
5
6# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -wao
# 结果如下
chr1 100 200 gene1 chr1 50 130 gene3 30
chr1 100 200 gene1 chr1 150 300 gene4 50
chr1 1000 1200 gene2 . -1 -1 . 0保留 A 的完整原始行,效果与-wa相同,但是去重
参数:-u (write original A entry once if any overlap)1
2
3
4# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -u
# 结果如下,效果与-wa相同,但是不再有重复行
chr1 100 200 gene1仅输出 A 中不与 B 重叠的区间
参数:-v (invert)1
2
3
4# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -v
# 结果如下
chr1 1000 1200 gene2基于重叠比例的过滤
参数:-f -F (minimum overlap fraction)1
2
3
4
5
6
7
8# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -f 0.5
# 结果如下,仅输出重叠部分占 A 区间长度 ≥50% 的区间
chr1 150 200 gene1
# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -F 0.35
# 结果如下,仅输出重叠部分占 B 区间长度 ≥35% 的区间
chr1 100 130 gene1统计 A 中每个区间与 B 的重叠次数(计数模式)
参数:-c (count overlaps)1
2
3
4
5# 执行命令
intersectBed -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -c
# 结果如下,输出 A 中的片段中
chr1 100 200 gene1 2
chr1 1000 1200 gene2 0
取并集
bedtools merge
bedtools merge 用于将重叠或相邻的基因组区间合并为连续的、非重叠的区间
合并重叠和连续的区间
1
2
3
4
5
6# 执行命令
bedtools merge -i <(echo -e "chr1\t50\t130\tgene3\nchr1\t100\t200\tgene1\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5\nchr1\t1000\t1200\tgene2")
# 结果如下
chr1 50 300
chr1 500 800
chr1 1000 1200允许合并的gap长度
参数:-d1
2
3
4# 执行命令
bedtools merge -i <(echo -e "chr1\t50\t130\tgene3\nchr1\t100\t200\tgene1\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5\nchr1\t1000\t1200\tgene2") -d 200
# 结果如下,合并间距小于等于200bp的片段为一个独立片段
chr1 50 1200合并时保留列信息
参数:-c -o1
2
3
4
5
6# 执行命令
bedtools merge -i <(echo -e "chr1\t50\t130\tgene3\nchr1\t100\t200\tgene1\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5\nchr1\t1000\t1200\tgene2") -c 4 -o distinct
# 保留了name列信息
chr1 50 300 gene1,gene3,gene4
chr1 500 800 gene5
chr1 1000 1200 gene2-c和-o需要联用。-o详细如下:类别 操作 示例 数学统计 sum,min,max,mean,median,stdev10+20 → 30 极值统计 absmin(最小绝对值),absmax,mode(众数),antimode(反众数)-5和3 → 3 (absmin) 列表操作 collapse(合并值),distinct(去重合并)“A,B,A”→A,B,A (collapse) 排序列表 distinct_sort_num(数值升序),distinct_sort_num_desc(降序)“3,1,2” → 1,2,3 计数 count(总数),count_distinct(唯一值数)“A,B,A” → 3 (count) 提取值 first,last“A,B,C” → A (first) 使用方法:
- 一对一模式:
-c 5,4 -o sum,distinct→ 第5列求和,第4列去重合并。 - 多对一模式:
-c 5,6 -o sum→ 两列均求和。 - 一对多模式:
-c 5 -o sum,mean→ 对第5列同时求和和求均值。
- 一对一模式:
链特异性合并
参数:-s1
2
3
4
5
6
7# 执行命令
bedtools merge -i <(echo -e "chr1\t50\t130\tgene3\t.\t+\nchr1\t100\t200\tgene1\t.\t+\nchr1\t150\t300\tgene4\t.\t-\nchr1\t500\t800\tgene5\t.\t+\nchr1\t1000\t1200\tgene2\t.\t+") -s -c 4,6 -o distinct
# 按照链方向合并区间
chr1 50 200 gene1,gene3 +
chr1 150 300 gene4 -
chr1 500 800 gene5 +
chr1 1000 1200 gene2 +
bedtools multiinter
可以同时分析多个 BED 文件重叠情况的工具,识别多个文件中共同出现的基因组区域,并统计每个区域在不同文件中的覆盖情况。
- 基本用法
1
2
3
4
5
6
7
8
9
10# 执行命令
bedtools multiinter -i <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5") -names A B
# 结果
chr1 50 100 1 B 0 1
chr1 100 130 2 A,B 1 1
chr1 130 150 1 A 1 0
chr1 150 200 2 A,B 1 1
chr1 200 300 1 B 0 1
chr1 500 800 1 B 0 1
chr1 1000 1200 1 A 1 0
取差集(bedtools subtract)
- 默认操作,从 A 中移除与 B 重叠的区域。
1
2
3
4
5# 执行命令
bedtools subtract -a <(echo -e "chr1\t100\t200\tgene1\nchr1\t1000\t1200\tgene2") -b <(echo -e "chr1\t50\t130\tgene3\nchr1\t150\t300\tgene4\nchr1\t500\t800\tgene5")
# 结果如下
chr1 130 150 gene1
chr1 1000 1200 gene2